Aviso: La página está en construcción.
Transformada de Fourier
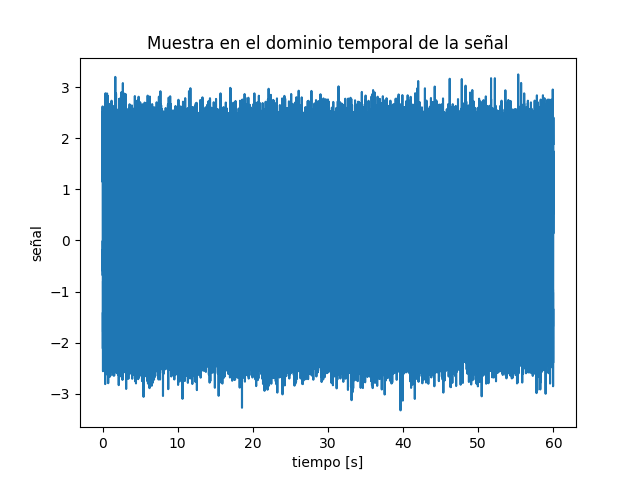
Ejemplo de datos frabricados:
from numpy import *
import matplotlib.pyplot as plt
random.seed(1024)
# Simulamos una frecuencia base de 10 Hz
f0 = 10 # Hz
# durante un tiempo de 60 segundos, con 100000 muestras equi-espaciadas
t = linspace(0,60,100000)
# Incluimos ruido de fase y ruido de amplitud
y1 = 2* cos(2*pi*f0*t + (random.rand(len(t))-0.5)/3 ) + random.randn(len(t))/3
# Y agregamos otra componente de 3.3 veces la frecuencia f0
y1 += ( 2* cos(2*pi*f0*t*3.3 + (random.rand(len(t))-0.5)/3 ) + random.randn(len(t))/3 ) /100
plt.figure(1)
plt.plot(t,y1)
plt.xlabel('tiempo [s]')
plt.ylabel('señal')
plt.title('Muestra en el dominio temporal de la señal')
# plt.savefig('03_01_simulacion_frecuencia.png')
from numpy import *
import matplotlib.pyplot as plt
# Para poder construir el eje de frecuencias de la transformada de Fourier hace
# falta:
# - Tener los datos en tiempos equi-espaciados
# - Saber el tiempo de separación entre los datos, que llamaremos t_step
t_step = mean(diff(t))
# Nos sirve saber el largo del vector temporal
N = len(y1)
ii = arange(N)
# Este factor de normalización corrige artificios de cálculo, debido a que
# la FFT no es un implementación completa de la Transformada de Fourier.
Norma = exp(-1j*ii[0:N//2]*pi/N) * 2/N
# Notar que, si no nos interesa la fase, lo único relevante de la Norma
# es el factor 2/N
Norma = 2/N
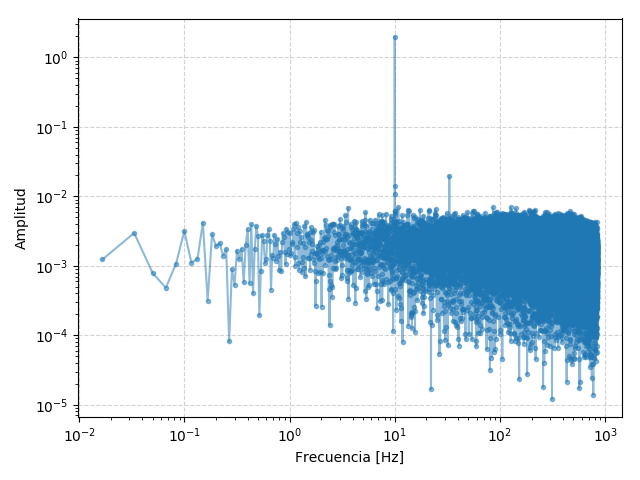
# Calculamos la transformada
Y1 = fft.rfft( y1 )[0:N//2] * Norma
ff = fft.fftfreq( N , d=t_step )[0:N//2]
# la expresión [0:N//2] es para quedarnos sólo con el semieje positivo de frecuencias
# Graficamos el valor absoluto en escala lograrítmica
plt.figure(2)
plt.plot( ff , abs(Y1) , '.-' , alpha=0.5 )
plt.semilogy()
plt.ylabel('Amplitud')
#plt.plot( ff , angle(Y1)*180/pi , *argv, **kwargs )
#plt.set_yticks([-180, -135, -90, -45, 0, 45, 90, 135, 180])
#plt.set_ylabel('fase [grad]')
plt.semilogx()
plt.xlabel('Frecuencia [Hz]')
plt.grid(True,linestyle='--',color='lightgray')
plt.tight_layout()
# plt.savefig('03_02_fft.png')
Filtros
Datos: espectro.npz
from numpy import *
import matplotlib.pyplot as plt
# Cargamos los datos
datos = load('espectro.npz')
tiempo = datos['tiempo']
medicion = datos['medicion']
# Y los graficamos
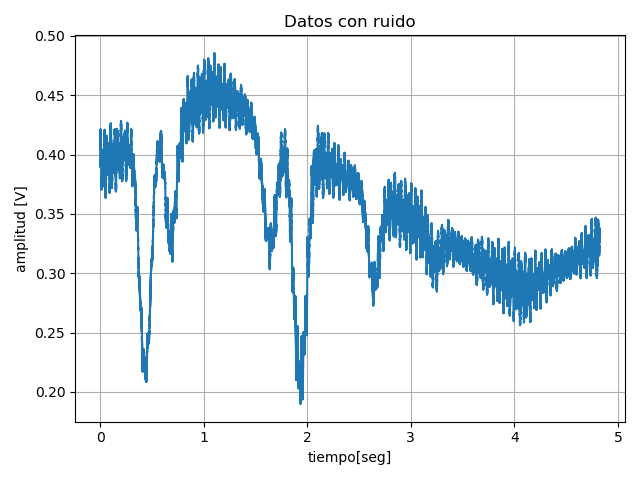
plt.figure(1)
plt.plot(tiempo , medicion )
plt.title('Datos con ruido')
plt.xlabel('tiempo[seg]')
plt.ylabel('amplitud [V]')
plt.grid(True)
plt.tight_layout()
# plt.savefig('03_03_datos_con_ruido.png')
# Para entender de donde puede venir el ruido, analizamos su espectro con FFT
plt.figure(2)
t = tiempo
y1 = medicion
t_step = mean(diff(t))
N = len(y1)
ii = arange(N)
Norma = exp(-1j*ii[0:N//2]*pi/N) * 2/N
Y1 = fft.rfft( y1 )[0:N//2] * Norma
ff = fft.fftfreq( N , d=t_step )[0:N//2]
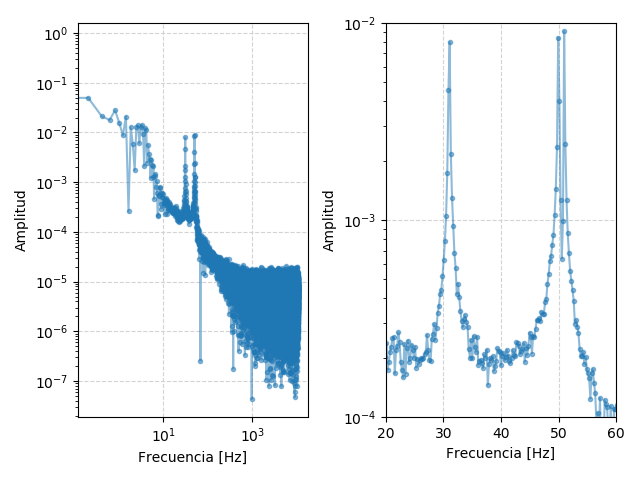
plt.subplot(1,2,1)
plt.plot( ff , abs(Y1) , '.-' , alpha=0.5 )
plt.semilogy()
plt.ylabel('Amplitud')
plt.semilogx()
plt.xlabel('Frecuencia [Hz]')
plt.grid(True,linestyle='--',color='lightgray')
plt.subplot(1,2,2)
plt.plot( ff , abs(Y1) , '.-' , alpha=0.5 )
plt.semilogy()
plt.ylabel('Amplitud')
#plt.semilogx()
plt.xlim(20,60)
plt.ylim(1e-4,1e-2)
plt.xlabel('Frecuencia [Hz]')
plt.grid(True,linestyle='--',color='lightgray')
plt.tight_layout()
# plt.savefig('03_04_datos_FFT.png')
from numpy import *
import matplotlib.pyplot as plt
from scipy.signal import savgol_filter as savgol
# La función savgol_filter permite implementar filtros de interpolación polinómica
#
# savgol( x , window_length , polyorder )
#
# savgol hace el siguiente procedimiento de aproximación:
# - Para cada punto toma una ventana de datos de largo: "window_length" (que siempre es impar)
# Por ejemplo, si window_length=7 , va a tomar los 3 datos anteriores y los 3 siguientes
# - En esa ventana va a ajustar los datos por un polinomio de orden: "polyorder"
# - Luego, devuelve el valor que tiene ese punto según esa función ajustada.
#
# Con esto podemos implementar varios tipos de filtros. Por ejemplo, el filtro de media movil.
# Esto es, reemplazar cada punto por el promedio de los N vecinos. En ese caso, es un
# polinomio de orden 0.
#
#
# Si queremos 10 vecinos (5 antes, 5 despues) sería aplicar:
# savgol( datos, 11 , 0 )
#
# Aplicamos filtros con promediado de 101 puntos y de 1001 puntos
print('Promedio de 101 pts equivale a: {:5.4f} segundos'.format( tiempo[101] -tiempo[0] ) )
print('Promedio de 1001 pts equivale a: {:5.4f} segundos'.format( tiempo[1001]-tiempo[0] ) )
# Promedio de 101 pts equivale a: 0.0049 segundos
# Promedio de 1001 pts equivale a: 0.0483 segundos
# En cada filtro ESTAMOS ELIMINANDO (atenunado en realidad) LA INFORMACIÓN
# DE TIEMPOS MAS RÁPIDOS QUE LA CANTIDAD DE SEGUNDOS MENCIONADA!!
plt.figure(4)
plt.subplot(1,2,1)
# Los datos para cada valor de filtro
plt.plot(tiempo , savgol(medicion,101,0) , label='101' , alpha=0.7 )
plt.plot(tiempo , savgol(medicion,1001,0) , label='1001' , alpha=0.7)
plt.plot(tiempo , savgol(savgol(medicion,1001,0),1001,0) , label='1001 dos veces' , linewidth=2)
plt.legend()
plt.grid(True)
plt.xlabel('tiempo[seg]')
plt.subplot(1,2,2)
# Lo mismo pero con zoom
plt.plot(tiempo , savgol(medicion,101,0) , label='101' , alpha=0.7 )
plt.plot(tiempo , savgol(medicion,1001,0) , label='1001' , alpha=0.7)
plt.plot(tiempo , savgol(savgol(medicion,1001,0),1001,0) , label='1001 dos veces' , linewidth=2)
# Referencia visual de los intervalos temporales de los filtros
plt.plot(tiempo[55899:56000] , 0.39*ones(101) , color='C0')
plt.plot(tiempo[55899:56900] , 0.385*ones(1001) , color='C1')
plt.plot(tiempo[55899:56900] , 0.38*ones(1001) , color='C2' , linewidth=2)
plt.xlim(2.65,3.1)
plt.ylim(0.3,0.4)
plt.xlabel('tiempo[seg]')
plt.grid(True)
plt.tight_layout()
# plt.savefig('03_05_smooth.png')
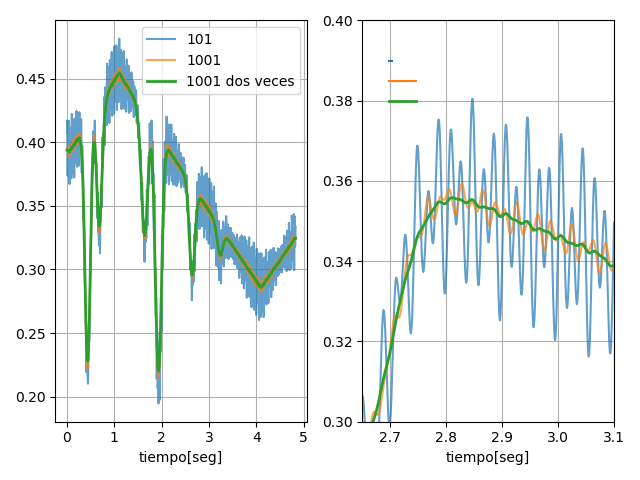
Comparación de los datos originales con dos series de datos filtrados.
from numpy import *
import matplotlib.pyplot as plt
from scipy.signal import savgol_filter as savgol
# Comparación de los datos filtrados con los originales
datos = load('espectro.npz')
tiempo = datos['tiempo']
medicion = datos['medicion']
# Y los graficamos
plt.figure(1)
filtro_A = savgol(savgol( medicion , 1001 , 0 ) , 1001, 0)
filtro_B = savgol( medicion , 3001 , 0 )
vectores = [medicion, filtro_A , filtro_B ]
nombres = ['Datos originales', 'Filtro A' , 'Filtro B' ]
# Dominio temporal ------------------------------------------------------------
plt.subplot(1,2,1)
for jj, vector in enumerate(vectores):
plt.plot(tiempo , vector , label=nombres[jj], alpha=0.7)
plt.title('Dominio Temporal')
plt.xlabel('tiempo [seg]')
plt.ylabel('amplitud [V]')
plt.grid(True,linestyle='--',color='lightgray')
plt.tight_layout()
plt.legend()
# Fourier ---------------------------------------------------------------------
plt.subplot(1,2,2)
for jj, vector in enumerate(vectores):
t = tiempo
y1 = vector
t_step = mean(diff(t))
N = len(y1)
ii = arange(N)
Norma = 2/N
Y1 = fft.rfft( y1 )[0:N//2] * Norma
ff = fft.fftfreq( N , d=t_step )[0:N//2]
plt.plot( ff , abs(Y1) , '-' , alpha=0.7 , label=nombres[jj])
plt.semilogy()
plt.ylabel('Amplitud')
#plt.semilogx()
plt.xlim(20,60)
plt.ylim(5e-5,1e-2)
plt.xlabel('Frecuencia [Hz]')
plt.title('Dominio de frecuencias')
plt.grid(True,linestyle='--',color='lightgray')
plt.tight_layout()
# plt.savefig('03_05_comparacion.png')
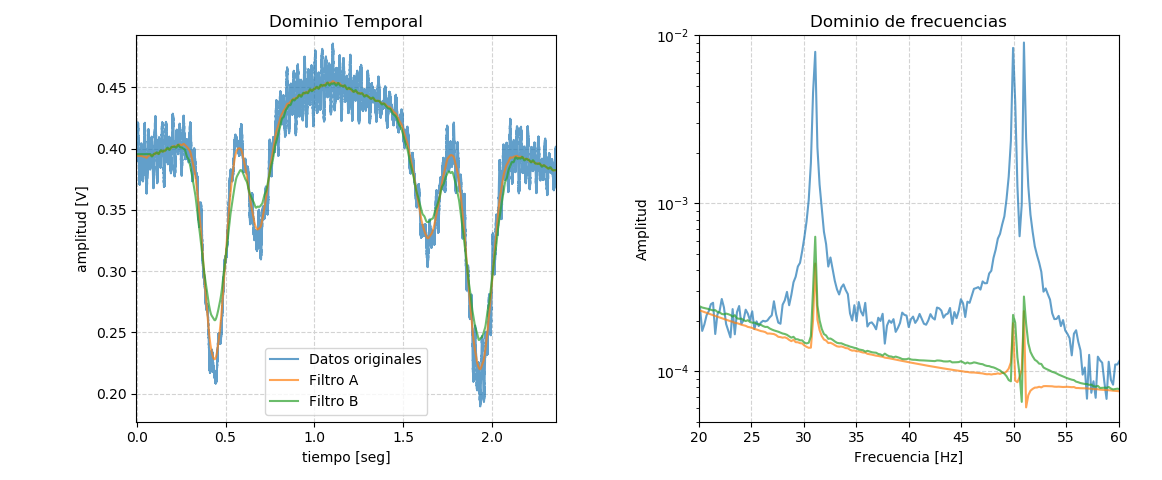
A continuación se muestra cómo utilizar scipy.signal.lfilter para simular filtros.
La diferencia cualitativa en estos casos es que los filtros operan como los filtros
en electrónica. Se muestra cómo implementar un filtro RC pasabajos.
En términos temporales, el savgol usa información del pasado y del futuro para
calcular los datos filtrados. En cambio, lfilter simula filtros físicos, donde
cada punto es construido solamente considerando la información del pasado.
from numpy import *
import matplotlib.pyplot as plt
from scipy.signal import butter, lfilter
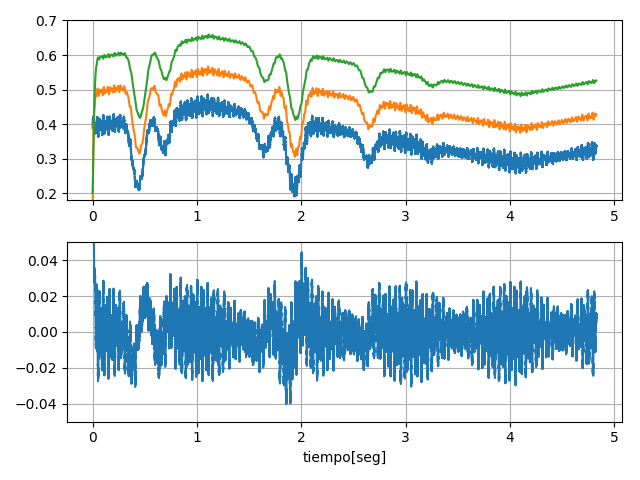
# Otra forma de filtrar los datos es simular la utilizacion de un filtro RC
# Ejemoplo de un filtro RC pasabajos con RC ~= 7.9 ms
# La frecuencia de corte es cutoff = 1/(2*pi*RC) = 20 Hz
fs = 1/mean(diff(tiempo))
cutoff = 20 # Hz
B, A = butter(1, cutoff / (fs / 2), btype='low') # 1st order Butterworth low-pass filter
medicion_low1 = lfilter(B, A, medicion , axis=0)
medicion_low2 = lfilter(B, A, medicion_low1 , axis=0)
# Ejemplo de filtro pasa-altos
B, A = butter(1, cutoff / (fs / 2), btype='high') # 1st order Butterworth low-pass filter
medicion_hig1 = lfilter(B, A, medicion , axis=0)
# Graficamos los datos con los diferentes filtros
# Agregamos un bias sólo para visualizarlo mejor
# Vemos el filtrado de frecuencia inferiores a 20 Hz y superiores a 20 Hz
plt.figure(5)
plt.subplot(2,1,1)
plt.plot(tiempo , 0 + medicion )
plt.plot(tiempo , 0.1+ medicion_low1 )
plt.plot(tiempo , 0.2+ medicion_low2 )
plt.grid(True)
plt.ylim(0.18,0.7)
#plt.xlabel('tiempo[seg]')
plt.subplot(2,1,2)
plt.plot(tiempo , medicion_hig1 )
plt.grid(True)
plt.xlabel('tiempo[seg]')
plt.ylim(-0.05,0.05)
plt.tight_layout()
# plt.savefig('03_06_filtro.png')
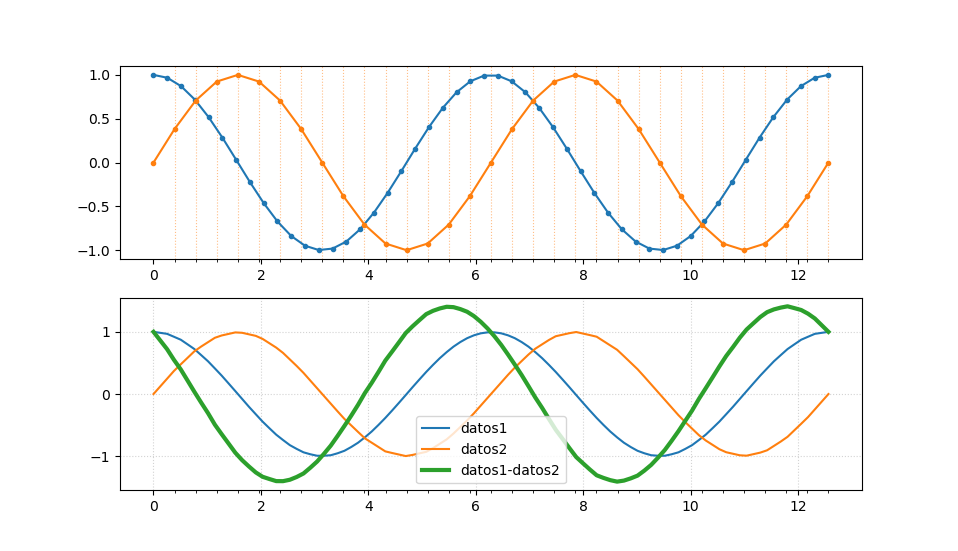
Interpolación
La función np.interp de NumPy permite obtener una base comun de tiempos para
realizar operaciones matemáticas entre datos adquiridos
# La interpolación sirve para poder hacer operaciones entre tiras de datos
# distintas que no comparten escalas de tiempos (entro otras cosas)
from numpy import *
import matplotlib.pyplot as plt
# Datos originales que no comparten base de tiempos
tiempo1 = linspace(0,4*pi, 50 )
datos1 = cos(tiempo1)
tiempo2 = linspace(0,4*pi, 33 )
datos2 = sin(tiempo2)
plt.figure()
# Graficamos los datos
plt.subplot(2,1,1)
plt.plot( tiempo1 , datos1 , '.-' )
plt.plot( tiempo2 , datos2 , '.-' )
# Señalamos las posiciones temporales de datos2
plt.gca().set_xticks( tiempo2 ,minor=True)
plt.grid(True,linestyle=':',color='C1', which='minor' , axis='x', alpha=0.5)
plt.subplot(2,1,2)
# Creamos nuevos vectores de datos que interpolando en forma lineal en las posiciones
# en que los valores no están definidos
tiempo_general = linspace(0,4*pi, 100 )
datos1_general = interp( tiempo_general , tiempo1, datos1 )
datos2_general = interp( tiempo_general , tiempo2, datos2 )
# Ahora que los vecotres tiene la misma base de tiempos, podemos hacer
# operaciones entre ellos
plt.plot( tiempo_general , datos1_general , '-' , label='datos1' )
plt.plot( tiempo_general , datos2_general , '-' , label='datos2' )
plt.plot( tiempo_general , datos1_general-datos2_general , '-' , label='datos1-datos2', lw=3 )
plt.gca().set_xticks( tiempo2 ,minor=True)
plt.grid(True,linestyle=':', color='lightgray')
plt.legend()
# plt.savefig('03_07_interp.png')
Análisis gráfico
Contenido pendiente